

const { OpenAI } = require('openai');
const api = new OpenAI({ apiKey: '', baseURL: 'https://api.ai.cc/v1' });
const main = async () => {
const prompt = `
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
`;
const response = await api.completions.create({
prompt,
model: 'togethercomputer/evo-1-131k-base',
});
const text = response.choices[0].text;
console.log('Completion:', text);
};
main();
from openai import OpenAI
client = OpenAI(
api_key="",
base_url="https://api.ai.cc/v1",
)
def main():
response = client.completions.create(
model="togethercomputer/evo-1-131k-base",
prompt="""
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
""",
)
completion = response.choices[0].text
print(f"Completion: {completion}")
main()

Produktdetails
✨ Evo-1 Basis (131K): Ein detaillierter Einblick
Evo-1 Base (131K) stellt einen bedeutenden Fortschritt im Bereich der KI-Modellierung dar und ist speziell auf die anspruchsvolle Verarbeitung von Texten und Genomsequenzen zugeschnitten. Dieses von Together Computer entwickelte Modell nutzt eine hochmoderne Architektur, um neben traditionellen Sprachverarbeitungsaufgaben eine beispiellose Leistung beim Verstehen und Generieren komplexer biologischer Daten zu erzielen.
Grundlegende Informationen
- ✨ Modellname: Evo-1 Basis (131K)
- 🚀 Entwickler/Schöpfer: Zusammen Computer
- 📅 Veröffentlichungsdatum: 25. Februar 2024
- ⚙️ Version: 1.1
- 💡 Modelltyp: Text-zu-Text-KI-Modell
Hauptmerkmale und Funktionen
Evo-1 Base (131K) basiert auf einer einzigartigen Architektur, die es ihm ermöglicht, komplexe Aufgaben mit umfangreichen Eingangsdaten zu bewältigen und ihn damit in der KI-Landschaft hervorzuheben.
- ✅ 7 Milliarden Parameter: Bietet umfangreiche Modellierungsmöglichkeiten für vielfältige Anwendungen.
- ✅ Architektur der Streifenhyäne: Verbessert die Effizienz der Sequenzverarbeitung, insbesondere bei langen Kontexten.
- ✅ Modellierung auf Einzelnukleotid-Ebene: Fähig zur präzisen Analyse genomischer Sequenzen.
- Trainiert auf OpenGenome: Profitiert von einem umfassenden Datensatz von ~300 Milliarden Token.
- ✅ Unterstützung für lange Kontexte: Verarbeitet Eingaben bis zu 131.000 Token, ideal für komplexe Aufgaben.
Bestimmungsgemäße Verwendung
Evo-1 wurde primär für Anwendungen in der Genomik, Bioinformatik und anderen Bereichen entwickelt, die eine hochauflösende Sequenzmodellierung erfordern. Seine Vielseitigkeit erstreckt sich aber auch auf allgemeine Sprachaufgaben.
- 🧬 Genomische Datenanalyse: Fortschrittliche DNA-Sequenzerzeugung und -analyse.
- ✍️ Inhaltsautomatisierung: Automatisierte Inhaltserstellung für verschiedene Bedürfnisse.
- 💬 Chatbot-Entwicklung: Entwicklung hochentwickelter Chatbots und Anwendungen zum Sprachverständnis.
- 🌐 Sprachaufgaben: Effiziente Sprachübersetzungs- und Zusammenfassungsaufgaben.
Sprachunterstützung
Das Modell unterstützt in erster Linie Englischsowie verschiedene biologische Sequenzformate für spezielle Anwendungen.
Technische Spezifikationen
Architektur
Evo-1 nutzt die Architektur der StreifenhyäneEs handelt sich um ein hybrides Design, das Multi-Head-Attention und Gated Convolutions kombiniert. Dieser innovative Ansatz ermöglicht die effiziente Verarbeitung außergewöhnlich langer Sequenzen und übertrifft herkömmliche Transformer-Modelle in bestimmten Kontexten.
Trainingsdaten
Das Modell wurde rigoros darauf trainiert. OpenGenome-Datensatz, eine umfangreiche Sammlung prokaryotischer Gesamtgenomsequenzen. Umfasst ungefähr 300 Milliarden TokenDieser Datensatz bietet eine solide Grundlage für das Erlernen komplexer biologischer Sequenzen. Dies steht im Gegensatz zu vielen Genommodellen, die oft mit kleineren, spezialisierten Datensätzen trainiert werden, was ihre Generalisierbarkeit einschränken kann.
Wissensgrenze
Die Wissensdatenbank von Evo-1 ist auf dem neuesten Stand (Stand: [Datum einfügen]). Februar 2024Die
Diversität und Voreingenommenheit
Die umfassende Natur der Trainingsdaten, die ein breites Spektrum prokaryotischer Genome abdecken, trägt wesentlich dazu bei, Verzerrungen zu reduzieren und die Fähigkeit des Modells zu verbessern, auf verschiedene biologische Kontexte zu generalisieren.
Leistungskennzahlen
Evo-1 Base (131K) hat in verschiedenen Benchmarks und spezialisierten Genomik-Aufgaben eine außergewöhnliche Leistung gezeigt:
- 📈 Genauigkeit: 89,5 % bei gängigen Textklassifizierungs-Benchmarks.
- 📊 Verwirrung: 8.3 auf dem Wikitext-103-Datensatz.
- ⭐ F1-Wertung: 92.7 zu Zusammenfassungsaufgaben.
- ⚡ Geschwindigkeit: Die Verarbeitung dauert pro Token etwa 12 ms und ermöglicht so Echtzeitanwendungen.
- 🛡️ Robustheit: Bewältigt mehrdeutige Anfragen und die Codegenerierung effizient und zeichnet sich durch bemerkenswerte Flexibilität aus.
Modernste Genomik-Fähigkeiten
- 🔬 Zero-Shot-Funktionsvorhersage: Bei der Vorhersage der Auswirkungen von Mutationen auf die Fitness konkurriert es mit spezialisierten domänenspezifischen Modellen und übertrifft diese oft.
- 🧪 Generierung mehrerer Elemente: Es zeichnet sich durch seine Fähigkeit aus, komplexe Molekülstrukturen zu erzeugen – eine neuartige Kompetenz für die synthetische Biologie.
- 🧬 Vorhersage der Genessenzialität: Ermöglicht präzise Vorhersagen der Genessenzialität auf Nukleotidebene, was für das Verständnis genetischer Funktionen von entscheidender Bedeutung ist.
Vergleich mit anderen Modellen

Das Evo-1 Base (131K)-Modell zeichnet sich als hochspezialisiertes Werkzeug aus für evolutionäre GenomanalyseWährend Modelle wie AlphaFold und RoseTTAFold führend in der Vorhersage von Proteinstrukturen sind, richtet sich Evo-1 Base speziell an Forscher, die sich auf groß angelegte Genomdaten konzentrieren, insbesondere zur Erforschung evolutionärer Muster und zur Erkennung von Mutationen über Arten hinweg.
Seine Effizienz bei der Verarbeitung umfangreicher Genomdatensätze macht es unverzichtbar für die Evolutionsbiologie, die vergleichende Genomik und die Mutationserkennung. Im Gegensatz zu proteinzentrierten Modellen wie ESM und ProtBert ist die Architektur von Evo-1 Base fein abgestimmt für Genomische Einblickeund positioniert es als eine leistungsstarke Option zur Förderung der Genomforschung und zum Verständnis der evolutionären Kräfte des Lebens.
Nutzung & Integration
Codebeispiele
Das Evo-1-Modell ist verfügbar auf KI/ML-API-Plattform unter der Kennung togethercomputer/evo-1-131k-baseDie
API-Dokumentation
Für umfassende Integrationsrichtlinien, detaillierte API-Dokumentation ist auf der Website der KI/ML-API verfügbar.
Ethische Richtlinien & Lizenzierung
Ethische Richtlinien
Die Entwicklung von Evo-1 hält sich strikt an ethische KI- und Bioinformatikstandardswobei der verantwortungsvolle Umgang mit Daten und die aktive Minimierung potenzieller Verzerrungen bei der Analyse genomischer Daten im Vordergrund stehen.
Lizenzierung
Das Modell wird unter der folgenden Bezeichnung veröffentlicht: Apache 2.0 Lizenz, wodurch weitreichende Rechte sowohl für die kommerzielle als auch für die nichtkommerzielle Nutzung gewährt werden.
❓ Häufig gestellte Fragen (FAQ)
F: Was macht Evo-1 Base (131K) für die Genomanalyse so einzigartig?
A: Evo-1 zeichnet sich durch seine StripedHyena-Architektur aus, die die Verarbeitung langer Kontexte (bis zu 131.000 Tokens) und das Training mit dem umfangreichen OpenGenome-Datensatz ermöglicht. Dies erlaubt eine beispiellose Auflösung bei der Modellierung genomischer Sequenzen, einschließlich Einzelnukleotid-Analysen und fortgeschrittener Aufgaben wie der Generierung mehrerer Elemente.
F: Kann Evo-1 Base für allgemeine Sprachaufgaben verwendet werden oder ist es nur für die Genomik geeignet?
A: Evo-1 Base ist zwar hochspezialisiert auf Genomik und Bioinformatik, aber dennoch ein vielseitiges KI-Modell für die Text-zu-Text-Verarbeitung. Es kann allgemeine Sprachaufgaben wie Inhaltsgenerierung, Zusammenfassung, Übersetzung und Chatbot-Entwicklung effektiv bewältigen und unterstützt dabei primär Englisch.
F: Welche Bedeutung hat die Architektur der Streifenhyäne?
A: Die StripedHyena-Architektur ist ein Hybrid-Design, das Multi-Head-Attention und Gated Convolutions kombiniert. Diese Innovation ermöglicht es Evo-1, lange Sequenzen effizienter zu verarbeiten als herkömmliche Transformer-Modelle. Dies ist entscheidend für komplexe Aufgaben, die umfangreiche Eingangsdaten erfordern, insbesondere in der Genomik.
F: Wie schneidet Evo-1 im Vergleich zu anderen prominenten KI-Modellen in der Biologie ab?
A: Evo-1 ist speziell auf die evolutionäre Genomanalyse und Mutationserkennung abgestimmt und unterscheidet sich dadurch von Modellen wie AlphaFold und RoseTTAFold (die sich auf die Vorhersage von Proteinstrukturen konzentrieren) oder ESM und ProtBert (optimiert für Proteinsequenzen). Die Stärke von Evo-1 liegt in der Interpretation umfangreicher Genomdaten und dem Verständnis evolutionärer Muster.
F: Unter welcher Lizenz wird Evo-1 Base (131K) veröffentlicht?
A: Evo-1 Base (131K) wird unter der Apache 2.0 Lizenz veröffentlicht, die sowohl die kommerzielle als auch die nichtkommerzielle Nutzung erlaubt und Entwicklern und Forschern somit große Flexibilität bietet.
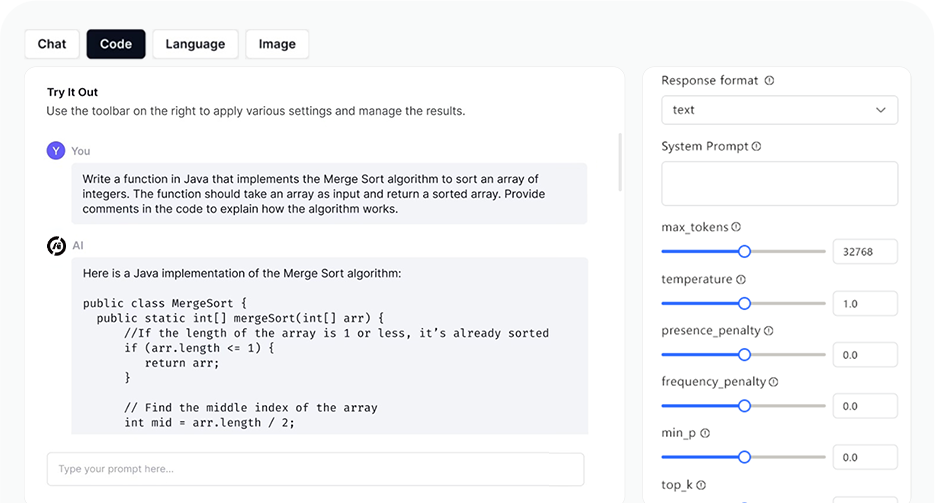
KI-Spielplatz


 Einloggen
Einloggen