

const { OpenAI } = require('openai');
const api = new OpenAI({ apiKey: '', baseURL: 'https://api.ai.cc/v1' });
const main = async () => {
const prompt = `
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
`;
const response = await api.completions.create({
prompt,
model: 'togethercomputer/evo-1-131k-base',
});
const text = response.choices[0].text;
console.log('Completion:', text);
};
main();
from openai import OpenAI
client = OpenAI(
api_key="",
base_url="https://api.ai.cc/v1",
)
def main():
response = client.completions.create(
model="togethercomputer/evo-1-131k-base",
prompt="""
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
""",
)
completion = response.choices[0].text
print(f"Completion: {completion}")
main()

Détails du produit
✨ Evo-1 Base (131K) : Analyse approfondie
Evo-1 Base (131K) représente une avancée majeure dans la modélisation par IA, spécifiquement conçue pour le traitement sophistiqué de textes et de séquences génomiques. Développé par Together Computer, ce modèle exploite une architecture de pointe pour offrir des performances inégalées dans la compréhension et la génération de données biologiques complexes, en plus des tâches linguistiques traditionnelles.
Informations de base
- ✨ Nom du modèle : Base Evo-1 (131K)
- 🚀 Développeur/Créateur : Ordinateur de groupe
- 📅 Date de sortie : 25 février 2024
- ⚙️ Version : 1.1
- 💡 Type de modèle : Modèle d'IA de texte à texte
Principales caractéristiques et capacités
Evo-1 Base (131K) est construit sur une architecture unique lui permettant de gérer des tâches complexes avec des données d'entrée volumineuses, ce qui le distingue dans le paysage de l'IA.
- ✅ 7 milliards de paramètres : Offre des capacités de modélisation étendues pour diverses applications.
- ✅ Architecture de StripedHyena : Améliore l'efficacité du traitement des séquences, notamment pour les contextes longs.
- ✅ Modélisation au niveau d'un seul nucléotide : Capable d'effectuer une analyse précise des séquences génomiques.
- Entraîné sur OpenGenome : Bénéficie d'un ensemble de données exhaustif d'environ 300 milliards de jetons.
- ✅ Prise en charge du contexte long : Traite jusqu'à 131 000 jetons en entrée, idéal pour les tâches complexes.
Utilisation prévue
Evo-1 est principalement conçu pour des applications en génomique, bioinformatique et autres domaines exigeant une modélisation de séquences à haute résolution. Sa polyvalence s'étend également aux tâches linguistiques générales.
- 🧬 Analyse des données génomiques : Génération et analyse avancées de séquences d'ADN.
- ✍️ Automatisation du contenu : Automatisation de la génération de contenu pour répondre à divers besoins.
- 💬 Développement de chatbots : Développement de chatbots sophistiqués et d'applications de compréhension du langage.
- 🌐 Tâches linguistiques : Tâches efficaces de traduction et de résumé linguistique.
Assistance linguistique
Le modèle prend principalement en charge Anglais, ainsi que divers formats de séquences biologiques pour des applications spécialisées.
Spécifications techniques
Architecture
Evo-1 utilise le Architecture de la hyène rayéeIl s'agit d'une conception hybride combinant l'attention multi-têtes et les convolutions à porte. Cette approche novatrice permet un traitement efficace des séquences exceptionnellement longues, surpassant les modèles de transformateurs traditionnels dans des contextes spécifiques.
Données d'entraînement
Le modèle a été rigoureusement entraîné sur le Ensemble de données OpenGenome, une vaste collection de séquences de génomes complets de procaryotes. Comprenant environ 300 milliards de jetonsCet ensemble de données offre une base solide pour l'apprentissage de séquences biologiques complexes. Cela contraste avec de nombreux modèles génomiques souvent entraînés sur des ensembles de données plus petits et spécialisés, ce qui peut limiter leur généralisation.
Seuil de connaissances
La base de connaissances d'Evo-1 est à jour au [date manquante]. Février 2024.
Diversité et préjugés
La nature exhaustive des données d'entraînement, couvrant un large éventail de génomes procaryotes, contribue significativement à réduire les biais et à améliorer la capacité du modèle à généraliser dans divers contextes biologiques.
Indicateurs de performance
Evo-1 Base (131K) a démontré des performances exceptionnelles sur divers benchmarks et tâches génomiques spécialisées :
- 📈 Précision : 89,5 % sur les tests de classification de texte courants.
- 📊 Perplexité : 8.3 sur l'ensemble de données Wikitext-103.
- ⭐ Score F1 : 92,7 sur les tâches de résumé.
- ⚡ Vitesse : Traite environ 12 ms par jeton, permettant des applications en temps réel.
- 🛡️ Robustesse : Gère efficacement les requêtes ambiguës et la génération de code, faisant preuve d'une flexibilité remarquable.
Capacités génomiques de pointe
- 🔬 Prédiction de fonction sans exemple : Il rivalise avec les modèles spécialisés et les surpasse souvent dans la prédiction des effets des mutations sur la forme physique.
- 🧪 Génération multi-éléments : Elle excelle dans la génération de structures moléculaires complexes, une capacité inédite pour la biologie synthétique.
- 🧬 Prédiction de l'essentialité des gènes : Prédit avec précision l'essentialité des gènes à la résolution du nucléotide, ce qui est crucial pour comprendre les fonctions génétiques.
Comparaison avec d'autres modèles

Le modèle Evo-1 Base (131K) se distingue comme un outil hautement spécialisé pour analyse génomique évolutiveAlors que des modèles comme AlphaFold et RoseTTAFold sont à la pointe de la prédiction de la structure des protéines, Evo-1 Base cible spécifiquement les chercheurs travaillant sur des données génomiques à grande échelle, notamment pour explorer les schémas évolutifs et détecter les mutations à travers les espèces.
Son efficacité avec de vastes ensembles de données génomiques la rend indispensable à la biologie évolutive, à la génomique comparative et à la détection de mutations. Contrairement aux modèles centrés sur les protéines tels que ESM et ProtBert, l'architecture d'Evo-1 Base est finement optimisée pour aperçus génomiques, ce qui en fait un choix judicieux pour faire progresser la recherche en génomique et comprendre les forces évolutives de la vie.
Utilisation et intégration
Exemples de code
Le modèle Evo-1 est accessible sur le Plateforme API IA/ML sous l'identifiant togethercomputer/evo-1-131k-base.
Documentation de l'API
Pour des directives d'intégration complètes et détaillées, consultez les détails Documentation de l'API est disponible sur le site web de l'API IA/ML.
Lignes directrices éthiques et agrément
Lignes directrices éthiques
Le développement de l'Evo-1 respecte strictement les normes éthiques en matière d'IA et de bioinformatique, en mettant l'accent sur une utilisation responsable et une minimisation active des biais potentiels dans l'analyse des données génomiques.
Licence
Le modèle est commercialisé sous la licence Licence Apache 2.0, accordant de larges droits pour une utilisation à la fois commerciale et non commerciale.
❓ Foire aux questions (FAQ)
Q : Qu'est-ce qui rend Evo-1 Base (131K) unique pour l'analyse génomique ?
A: Evo-1 se distingue par son architecture StripedHyena, permettant le traitement de contextes longs (jusqu'à 131 000 jetons) et l'entraînement sur le vaste ensemble de données OpenGenome. Ceci permet une résolution inégalée dans la modélisation des séquences génomiques, y compris l'analyse au niveau du nucléotide unique et des tâches avancées comme la génération multi-éléments.
Q : La base Evo-1 peut-elle être utilisée pour des tâches linguistiques générales, ou est-elle uniquement destinée à la génomique ?
A: Bien qu'Evo-1 Base soit hautement spécialisé en génomique et bioinformatique, il s'agit d'un modèle d'IA polyvalent pour le traitement de texte. Il peut gérer efficacement des tâches linguistiques générales telles que la génération de contenu, la synthèse, la traduction et le développement de chatbots, principalement en anglais.
Q : Quelle est la signification de l'architecture de la hyène rayée ?
A : L'architecture StripedHyena est une conception hybride combinant l'attention multi-têtes et les convolutions à porte. Cette innovation permet à Evo-1 de traiter les longues séquences plus efficacement que les modèles de transformateurs traditionnels, ce qui est crucial pour les tâches complexes nécessitant d'importantes quantités de données d'entrée, notamment en génomique.
Q : Comment Evo-1 se compare-t-il aux autres modèles d'IA importants en biologie ?
A: Evo-1 est spécifiquement conçu pour l'analyse génomique évolutive et la détection des mutations, ce qui le distingue de modèles comme AlphaFold et RoseTTAFold (axés sur la prédiction de la structure des protéines) ou ESM et ProtBert (optimisés pour les séquences protéiques). La force d'Evo-1 réside dans l'interprétation de données génomiques à grande échelle et la compréhension des schémas évolutifs.
Q : Sous quelle licence Evo-1 Base (131K) est-il distribué ?
A: Evo-1 Base (131K) est distribué sous la licence Apache 2.0, qui autorise une utilisation commerciale et non commerciale, offrant une grande flexibilité aux développeurs et aux chercheurs.
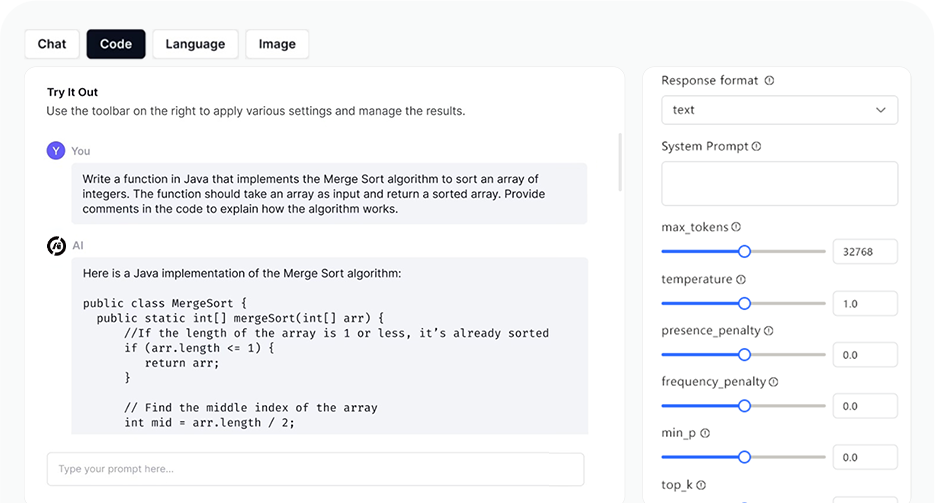
Terrain de jeu de l'IA


 Se connecter
Se connecter