

const { OpenAI } = require('openai');
const api = new OpenAI({ apiKey: '', baseURL: 'https://api.ai.cc/v1' });
const main = async () => {
const prompt = `
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
`;
const response = await api.completions.create({
prompt,
model: 'togethercomputer/evo-1-131k-base',
});
const text = response.choices[0].text;
console.log('Completion:', text);
};
main();
from openai import OpenAI
client = OpenAI(
api_key="",
base_url="https://api.ai.cc/v1",
)
def main():
response = client.completions.create(
model="togethercomputer/evo-1-131k-base",
prompt="""
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
""",
)
completion = response.choices[0].text
print(f"Completion: {completion}")
main()

Detalle del producto
✨ Base Evo-1 (131K): Un análisis profundo
Evo-1 Base (131K) representa un avance significativo en el modelado de IA, específicamente diseñado para el procesamiento sofisticado de texto y secuencias genómicas. Desarrollado por Together Computer, este modelo aprovecha una arquitectura de vanguardia para ofrecer un rendimiento inigualable en la comprensión y generación de datos biológicos complejos, junto con las tareas lingüísticas tradicionales.
Información básica
- ✨ Nombre del modelo: Base Evo-1 (131K)
- 🚀 Desarrollador/Creador: Juntos Computadora
- 📅 Fecha de lanzamiento: 25 de febrero de 2024
- ⚙️ Versión: 1.1
- 💡Tipo de modelo: Modelo de IA de texto a texto
Características y capacidades clave
Evo-1 Base (131K) está construido sobre una arquitectura única que le permite manejar tareas complejas con amplios datos de entrada, lo que lo distingue en el panorama de la IA.
- ✅ 7 mil millones de parámetros: Proporciona amplias capacidades de modelado para diversas aplicaciones.
- ✅ Arquitectura de la hiena rayada: Mejora la eficiencia del procesamiento de secuencias, especialmente para contextos largos.
- ✅ Modelado a nivel de un solo nucleótido: Capaz de análisis preciso de secuencias genómicas.
- Capacitado en OpenGenome: Se beneficia de un conjunto de datos completo de ~300 mil millones de tokens.
- ✅ Soporte de contexto largo: Procesa entradas de hasta 131K tokens, ideal para tareas complejas.
Uso previsto
Evo-1 está diseñado principalmente para aplicaciones en genómica, bioinformática y otros campos que exigen modelado de secuencias de alta resolución. Su versatilidad también se extiende a tareas lingüísticas generales.
- 🧬 Análisis de datos genómicos: Generación y análisis avanzado de secuencias de ADN.
- ✍️ Automatización de contenido: Automatizar la generación de contenidos para diversas necesidades.
- 💬 Desarrollo de Chatbot: Construcción de chatbots sofisticados y aplicaciones de comprensión del lenguaje.
- 🌐 Tareas de idioma: Tareas eficientes de traducción y resumen de idiomas.
Soporte de idiomas
El modelo apoya principalmente Inglés, junto con varios formatos de secuencias biológicas para aplicaciones especializadas.
Especificaciones técnicas
Arquitectura
Evo-1 utiliza el Arquitectura de la hiena rayadaUn diseño híbrido que combina la atención multicabezal y convoluciones controladas. Este innovador enfoque permite el procesamiento eficiente de secuencias excepcionalmente largas, superando a los modelos de transformador tradicionales en contextos específicos.
Datos de entrenamiento
El modelo fue entrenado rigurosamente en el Conjunto de datos OpenGenome, una extensa colección de secuencias del genoma completo de procariotas. Comprende aproximadamente 300 mil millones de tokensEste conjunto de datos proporciona una base sólida para el aprendizaje de secuencias biológicas complejas. Esto contrasta con muchos modelos genómicos que suelen entrenarse con conjuntos de datos más pequeños y especializados, lo que puede limitar su generalización.
Límite de conocimiento
La base de conocimientos de Evo-1 está actualizada a partir de Febrero de 2024.
Diversidad y sesgo
La naturaleza integral de los datos de entrenamiento, que abarcan una amplia gama de genomas procariotas, ayuda significativamente a reducir el sesgo y mejorar la capacidad del modelo para generalizarse en diversos contextos biológicos.
Métricas de rendimiento
Evo-1 Base (131K) ha demostrado un rendimiento excepcional en varios puntos de referencia y tareas genómicas especializadas:
- 📈 Precisión: 89,5% en puntos de referencia de clasificación de texto comunes.
- 📊 Perplejidad: 8.3 en el conjunto de datos Wikitext-103.
- ⭐ Puntuación F1: 92.7 en tareas de resumen.
- ⚡Velocidad: Procesa aproximadamente 12 ms por token, lo que permite aplicaciones en tiempo real.
- 🛡️ Robustez: Maneja eficientemente consultas ambiguas y generación de código, mostrando una notable flexibilidad.
Capacidades genómicas de vanguardia
- 🔬 Predicción de la función de disparo cero: Compite con, y a menudo supera, los modelos especializados de dominios específicos a la hora de predecir los efectos de las mutaciones sobre la aptitud.
- 🧪 Generación de múltiples elementos: Se destaca en la generación de estructuras moleculares complejas, una capacidad novedosa para la biología sintética.
- 🧬 Predicción de esencialidad genética: Predice con precisión la esencialidad de los genes en la resolución de nucleótidos, crucial para comprender las funciones genéticas.
Comparación con otros modelos

El modelo Evo-1 Base (131K) se distingue por ser una herramienta altamente especializada para análisis genómico evolutivoSi bien modelos como AlphaFold y RoseTTAFold son líderes en la predicción de la estructura de proteínas, Evo-1 Base está dirigido exclusivamente a investigadores centrados en datos genómicos a gran escala, en particular para explorar patrones evolutivos y detectar mutaciones entre especies.
Su eficiencia con vastos conjuntos de datos genómicos la hace indispensable para la biología evolutiva, la genómica comparativa y la detección de mutaciones. A diferencia de los modelos centrados en proteínas como ESM y ProtBert, la arquitectura de Evo-1 Base está optimizada para conocimientos genómicos, posicionándolo como una opción poderosa para avanzar en la investigación en genómica y comprender las fuerzas evolutivas de la vida.
Uso e integración
Ejemplos de código
El modelo Evo-1 está accesible en el Plataforma API de IA/ML bajo el identificador juntoscomputadora/evo-1-131k-base.
Documentación de la API
Para obtener pautas de integración completas y detalladas Documentación de la API Está disponible en el sitio web de la API de IA/ML.
Pautas éticas y licencias
Directrices éticas
El desarrollo de Evo-1 se adhiere estrictamente a Estándares éticos de IA y bioinformática, haciendo hincapié en el uso responsable y la minimización activa de posibles sesgos en el análisis de datos genómicos.
Licencias
El modelo se lanza bajo la Licencia Apache 2.0, otorgando amplios derechos tanto para uso comercial como no comercial.
❓ Preguntas frecuentes (FAQ)
P: ¿Qué hace que Evo-1 Base (131K) sea único para el análisis genómico?
R: Evo-1 destaca por su arquitectura StripedHyena, que permite el procesamiento de contextos extensos (hasta 131 000 tokens) y el entrenamiento en el vasto conjunto de datos OpenGenome. Esto permite una resolución inigualable en el modelado de secuencias genómicas, incluyendo el análisis a nivel de un solo nucleótido y tareas avanzadas como la generación de múltiples elementos.
P: ¿Se puede utilizar Evo-1 Base para tareas de lenguaje general o es solo para genómica?
R: Si bien está altamente especializado en genómica y bioinformática, Evo-1 Base es un modelo de IA texto a texto versátil. Puede gestionar eficazmente tareas lingüísticas generales, como la generación de contenido, la síntesis, la traducción y el desarrollo de chatbots, principalmente en inglés.
P: ¿Cuál es el significado de la arquitectura de StripedHyena?
R: La arquitectura de StripedHyena es un diseño híbrido que combina la atención multicabezal y convoluciones controladas. Esta innovación permite a Evo-1 procesar secuencias largas con mayor eficiencia que los modelos de transformador tradicionales, lo cual es crucial para tareas complejas que requieren una gran cantidad de datos de entrada, especialmente en genómica.
P: ¿Cómo se compara Evo-1 con otros modelos de IA destacados en biología?
R: Evo-1 está optimizado para el análisis genómico evolutivo y la detección de mutaciones, lo que lo diferencia de modelos como AlphaFold y RoseTTAFold (centrados en la predicción de la estructura de proteínas) o ESM y ProtBert (optimizados para secuencias de proteínas). La fortaleza de Evo-1 reside en la interpretación de datos genómicos a gran escala y la comprensión de patrones evolutivos.
P: ¿Bajo qué licencia se lanzó Evo-1 Base (131K)?
A: Evo-1 Base (131K) se publica bajo la licencia Apache 2.0, que permite el uso comercial y no comercial y ofrece una amplia flexibilidad para desarrolladores e investigadores.
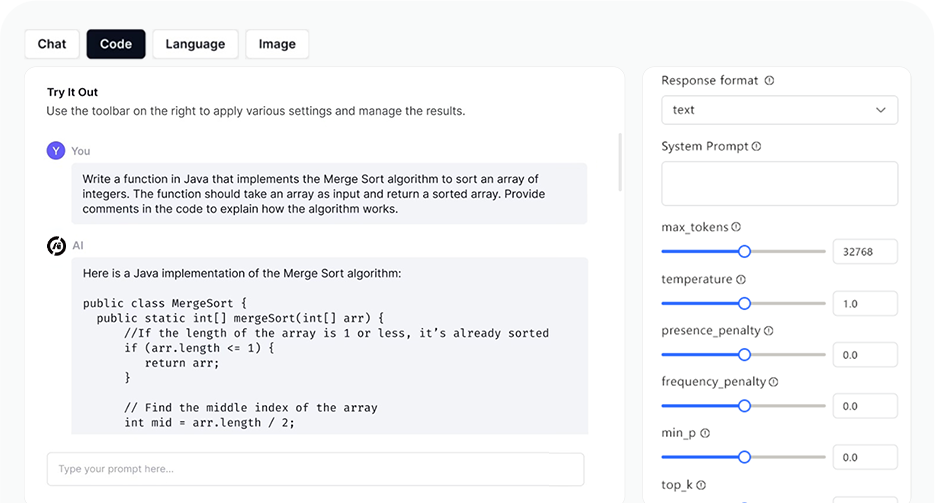
Patio de juegos de IA


 Acceso
Acceso