

const { OpenAI } = require('openai');
const api = new OpenAI({ apiKey: '', baseURL: 'https://api.ai.cc/v1' });
const main = async () => {
const prompt = `
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
`;
const response = await api.completions.create({
prompt,
model: 'togethercomputer/evo-1-131k-base',
});
const text = response.choices[0].text;
console.log('Completion:', text);
};
main();
from openai import OpenAI
client = OpenAI(
api_key="",
base_url="https://api.ai.cc/v1",
)
def main():
response = client.completions.create(
model="togethercomputer/evo-1-131k-base",
prompt="""
All of the states in the USA:
- Alabama, Mongomery;
- Arkansas, Little Rock;
""",
)
completion = response.choices[0].text
print(f"Completion: {completion}")
main()

Detalhes do produto
✨ Base Evo-1 (131K): Uma Análise Detalhada
O Evo-1 Base (131K) representa um avanço significativo na modelagem de IA, especificamente desenvolvido para o processamento sofisticado de textos e sequências genômicas. Desenvolvido pela Together Computer, este modelo utiliza arquitetura de ponta para oferecer desempenho incomparável na compreensão e geração de dados biológicos complexos, além de tarefas tradicionais de linguagem.
Informações básicas
- ✨ Nome do modelo: Base Evo-1 (131K)
- 🚀 Desenvolvedor/Criador: Computadores Juntos
- 📅 Data de lançamento: 25 de fevereiro de 2024
- ⚙️ Versão: 1.1
- 💡 Tipo de modelo: Modelo de IA de texto para texto
Principais características e funcionalidades
O Evo-1 Base (131K) é construído sobre uma arquitetura única que lhe permite lidar com tarefas complexas com grandes volumes de dados de entrada, diferenciando-o no cenário da IA.
- ✅ 7 bilhões de parâmetros: Oferece amplas capacidades de modelagem para diversas aplicações.
- ✅ Arquitetura StripedHyena: Aumenta a eficiência do processamento de sequências, especialmente em contextos longos.
- ✅ Modelagem em nível de nucleotídeo único: Capaz de realizar análises precisas de sequências genômicas.
- Treinado no OpenGenome: Beneficia-se de um conjunto de dados abrangente com cerca de 300 bilhões de tokens.
- ✅ Suporte para contextos longos: Processa entradas de até 131 mil tokens, ideal para tarefas complexas.
Uso pretendido
O Evo-1 foi projetado principalmente para aplicações em genômica, bioinformática e outras áreas que exigem modelagem de sequências de alta resolução. Sua versatilidade também se estende a tarefas gerais de linguagem.
- 🧬 Análise de Dados Genômicos: Geração e análise avançadas de sequências de DNA.
- ✍️ Automação de conteúdo: Automatizar a geração de conteúdo para diversas necessidades.
- 💬 Desenvolvimento de chatbots: Desenvolvimento de chatbots sofisticados e aplicativos de compreensão de linguagem natural.
- 🌐 Tarefas de linguagem: Tarefas eficientes de tradução e sumarização de idiomas.
Suporte linguístico
O modelo apoia principalmente Inglês, juntamente com vários formatos de sequências biológicas para aplicações especializadas.
Especificações técnicas
Arquitetura
O Evo-1 utiliza o Arquitetura da Hiena Listrada, um design híbrido que combina atenção multi-cabeças e convoluções controladas. Essa abordagem inovadora permite o processamento eficiente de sequências excepcionalmente longas, superando os modelos Transformer tradicionais em contextos específicos.
Dados de treinamento
O modelo foi rigorosamente treinado no Conjunto de dados OpenGenome, uma extensa coleção de sequências de genomas completos de procariontes. Compreendendo aproximadamente 300 bilhões de tokensEste conjunto de dados fornece uma base sólida para o aprendizado de sequências biológicas complexas. Isso contrasta com muitos modelos genômicos frequentemente treinados em conjuntos de dados menores e especializados, o que pode limitar sua generalização.
Limite de conhecimento
A base de conhecimento do Evo-1 está atualizada até a data de Fevereiro de 2024.
Diversidade e Viés
A abrangência dos dados de treinamento, que cobrem uma ampla gama de genomas procarióticos, contribui significativamente para reduzir o viés e aprimorar a capacidade do modelo de generalizar em diversos contextos biológicos.
Métricas de desempenho
A Evo-1 Base (131K) demonstrou desempenho excepcional em diversos benchmarks e tarefas genômicas especializadas:
- 📈 Precisão: 89,5% em benchmarks comuns de classificação de texto.
- 📊 Perplexidade: 8.3 no conjunto de dados Wikitext-103.
- ⭐ Pontuação F1: 92,7 em tarefas de sumarização.
- ⚡ Velocidade: Processa aproximadamente 12ms por token, permitindo aplicações em tempo real.
- 🛡️ Robustez: Lida de forma eficiente com consultas ambíguas e geração de código, demonstrando notável flexibilidade.
Capacidades genômicas de ponta
- 🔬 Previsão de função sem exemplos (Zero-shot Function Prediction): Compete com modelos especializados e frequentemente os supera na previsão dos efeitos das mutações na aptidão.
- 🧪 Geração de múltiplos elementos: Destaca-se na geração de estruturas moleculares complexas, uma capacidade inovadora para a biologia sintética.
- 🧬 Previsão de Essencialidade Genética: Prevê com precisão a essencialidade dos genes em resolução de nucleotídeos, o que é crucial para a compreensão das funções genéticas.
Comparação com outros modelos

O modelo Evo-1 Base (131K) se destaca como uma ferramenta altamente especializada para análise genômica evolutivaEmbora modelos como AlphaFold e RoseTTAFold sejam líderes na predição da estrutura de proteínas, o Evo-1 Base se destina exclusivamente a pesquisadores focados em dados genômicos em larga escala, particularmente para explorar padrões evolutivos e detectar mutações entre espécies.
Sua eficiência com vastos conjuntos de dados genômicos o torna indispensável para biologia evolutiva, genômica comparativa e detecção de mutações. Ao contrário de modelos centrados em proteínas, como ESM e ProtBert, a arquitetura do Evo-1 Base é finamente ajustada para insights genômicos, posicionando-a como uma escolha poderosa para o avanço da pesquisa em genômica e para a compreensão das forças evolutivas da vida.
Utilização e integração
Exemplos de código
O modelo Evo-1 está disponível em Plataforma de API de IA/ML sob o identificador togethercomputer/evo-1-131k-base.
Documentação da API
Para obter diretrizes de integração abrangentes e detalhadas, consulte os detalhes. Documentação da API Está disponível no site da API de IA/ML.
Diretrizes Éticas e Licenciamento
Diretrizes Éticas
O desenvolvimento do Evo-1 segue rigorosamente as diretrizes. padrões éticos de IA e bioinformática, enfatizando o uso responsável e a minimização ativa de potenciais vieses na análise de dados genômicos.
Licenciamento
O modelo é lançado sob a Licença Apache 2.0, concedendo amplos direitos tanto para uso comercial quanto não comercial.
❓ Perguntas frequentes (FAQ)
P: O que torna a base Evo-1 (131K) única para análise genômica?
A: O Evo-1 se destaca devido à sua arquitetura StripedHyena, que permite o processamento de contextos longos (até 131 mil tokens) e o treinamento no vasto conjunto de dados OpenGenome. Isso possibilita uma resolução incomparável na modelagem de sequências genômicas, incluindo análises em nível de nucleotídeo único e tarefas avançadas como a geração de múltiplos elementos.
P: O Evo-1 Base pode ser usado para tarefas gerais de linguagem ou é exclusivo para genômica?
A: Embora altamente especializado em genômica e bioinformática, o Evo-1 Base é um modelo de IA versátil para conversão de texto em texto. Ele pode lidar eficazmente com tarefas gerais de linguagem, como geração de conteúdo, sumarização, tradução e desenvolvimento de chatbots, com suporte principalmente para o inglês.
P: Qual é o significado da arquitetura da Hiena-listrada?
A: A arquitetura StripedHyena é um design híbrido que combina atenção multi-cabeças e convoluções controladas. Essa inovação permite que o Evo-1 processe sequências longas com mais eficiência do que os modelos Transformer tradicionais, o que é crucial para tarefas complexas que exigem grandes quantidades de dados de entrada, principalmente em genômica.
P: Como o Evo-1 se compara a outros modelos de IA proeminentes na biologia?
A: O Evo-1 é otimizado exclusivamente para análise genômica evolutiva e detecção de mutações, diferenciando-se de modelos como AlphaFold e RoseTTAFold (focados na predição da estrutura de proteínas) ou ESM e ProtBert (otimizados para sequências de proteínas). A principal vantagem do Evo-1 reside na interpretação de dados genômicos em larga escala e na compreensão de padrões evolutivos.
P: Sob qual licença o Evo-1 Base (131K) foi lançado?
A: Evo-1 Base (131K) é distribuído sob a licença Apache 2.0, que permite o uso comercial e não comercial, oferecendo ampla flexibilidade para desenvolvedores e pesquisadores.
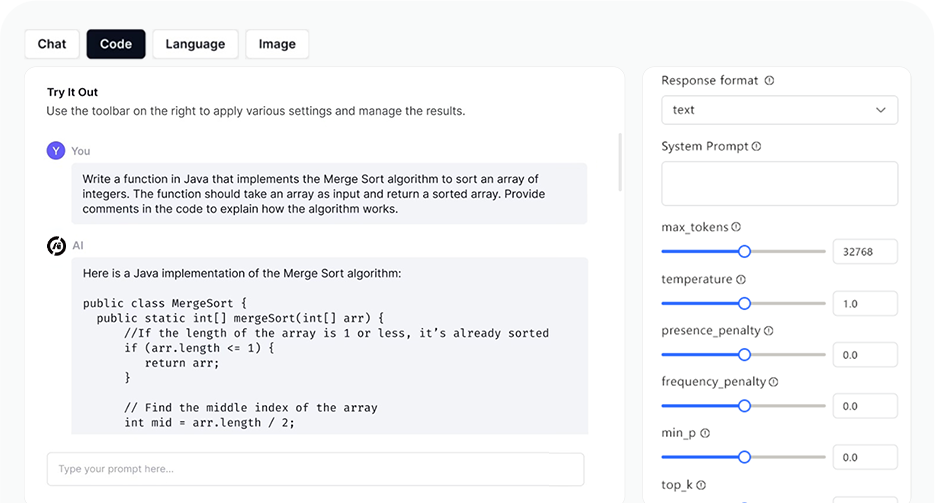
Playground de IA


 Conecte-se
Conecte-se